近日,我校郑春厚/苏延森教授团队在DNA调控元件智能预测与设计方面取得新进展。相关成果作为 封面文章发表于生物信息学领域顶级期刊 Nucleic Acids Research(DOI:10.1093/nar/gkaf833)。
启动子是控制基因表达的关键功能元件,设计和优化新的功能启动子对于代谢通路改造和基因治疗具有重要意义。传统实验方法依赖突变或片段重组,耗时耗力且无法覆盖整个序列空间。基于深度学习的方法在合成启动子任务中展示出巨大的潜力,但现有研究大多集中在如何设计强启动子,忽略实际应用中对不同表达强度启动子的需求。针对上述问题,团队提出一种全新的启动子设计方法PromoDGDE,结合深度生成模型和进化算法,实现对启动子序列的从头设计和按需优化,并将其应用于大肠杆菌和酿酒酵母启动子设计任务。生物实验结果表明,所有合成启动子均具有活性,且超过60%的序列显示出预期的调控水平。该研究为人工智能技术赋能合成生物学提供了新的技术支撑。
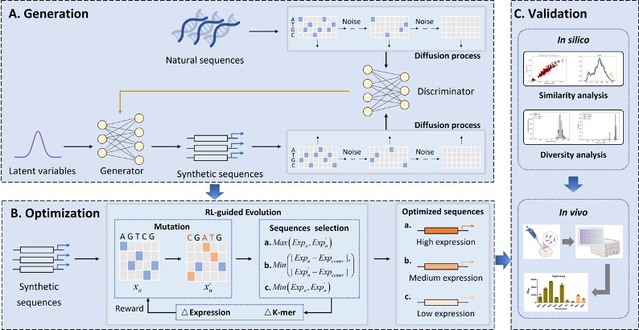
图1 基于深度生成模型和进化算法的启动子设计框架

图2 期刊封面






 1476
1476 电子营业执照
电子营业执照